主成分分析 (Principal Component Analysis, PCA) は、1観測あたり多数の次元/特徴を含む大規模データセットを分析し、最大限の情報量を保持しながらデータの解釈可能性を高め、多次元データの可視化を可能にする手法としてよく利用されています。形式的には、PCA はデータセットの次元を減少させる統計的手法です。これは、データの変動(の大部分)が、初期データよりも少ない次元で記述できるような新しい座標系にデータを線形変換することにより達成できます。
データ解析で頻繁に主成分分析 (PCA) を利用しています。主成分分析をはじめ、よく使用する機械学習の処理部分などは、いつも過去に使ったスクリプトから必要部分をコピペして使い回してしまっているので、思い違いや間違いがあれば、そのままずっと引きずってしまう可能性があります。ちょっと心配になってきたので、簡単なサンプルを使ってスクリプトを整理しようとしています。
まずは主成分分析
今回の目的は、主成分分析で変換された主成分それぞれが、ターゲットに対してどの程度影響があるかを、一元配置分散分析(以降、単に分散分析と表します)を利用して検定することです。
下記の OS 環境で動作確認をしています。

|
Fedora Linux 37 Workstation |
x86_64 |
| Python |
3.10.9 |
JupyterLab の Notebook 上で動作確認をしています。今回も主成分分析の結果を利用しますので、まず、主成分分析をまとめて処理してしまいます。本ブログ記事 [1] で紹介した Iris データセットの主成分分析の処理を、必要な部分だけぎゅっとひとつにまとめました。
import pandas as pd
import dataframe_image as dfi
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
# https://www.kaggle.com/datasets/saurabh00007/iriscsv
filename = 'Iris.csv'
df = pd.read_csv(filename, index_col=0)
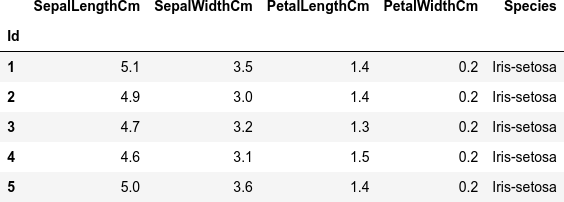
dfi.export(df.head(), 'table_041_iris.png')
print(df.head())
cols_x = list(df.columns[0:4])
col_y = df.columns[4]
# model pipeline for PCA
pipe = Pipeline(steps=[
('scaler', StandardScaler()),
('PCA', PCA()),
])
features = df[cols_x]
pipe.fit(features)
# PCA scores
scores = pipe.transform(features)
df_pca = pd.DataFrame(
scores,
columns=["PC{}".format(x + 1) for x in range(scores.shape[1])],
index=df.index
)
cols_pc = list(df_pca.columns)
df_pca.insert(0, col_y, df[col_y].copy())
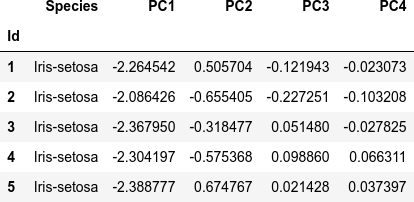
dfi.export(df_pca.head(), 'table_041_iris_PCA.png')
print(df_pca.head())
これが、読み込んだ Iris のデータセット df(最初の5行)です。
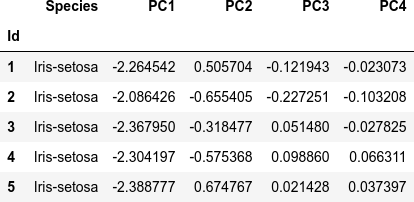
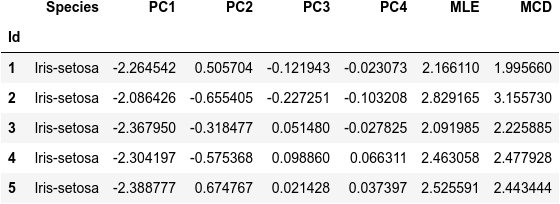
次が主成分分析をしたデータセット df_pca(最初の5行)です。
生データの分散分析
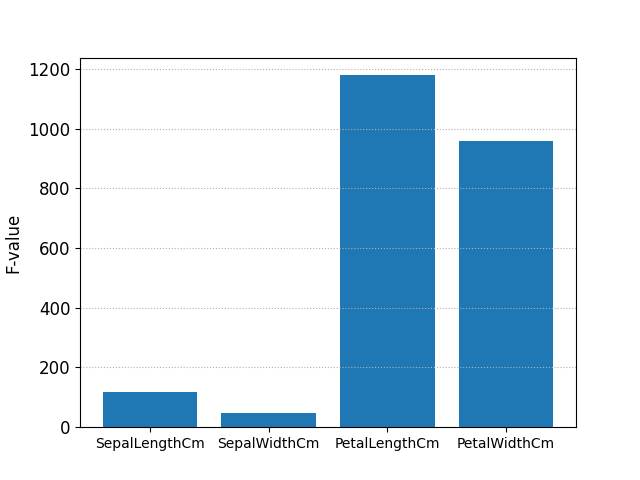
分散分析は SciPy の stats ライブラリの f_oneway を利用します。個々の因子 (SepalLengthCm, SepalWidthCm, PetalLengthCm, PetalWidthCm) に対し、ターゲット (Species) それぞれ (Iris-setosa, Iris-versicolor, Iris-virginica) の群をリストにまとめて、f_oneway() に渡します。
import numpy as np
from scipy import stats
list_species = list(set(df_pca[col_y]))
cols_stat = ['F-value', 'p-value']
df_anova = pd.DataFrame(columns=cols_stat)
for x in cols_x:
args = [df[df[col_y] == list_species[i]][x] for i, species in enumerate(list_species)]
f_value, p_value = stats.f_oneway(*args)
result = pd.DataFrame(
np.array([f_value, p_value]).reshape(1, 2),
columns=cols_stat,
index=[x]
)
# append row
df_anova = pd.concat([df_anova, result], axis=0)
print(df_anova)
F-value p-value
SepalLengthCm 119.264502 1.669669e-31
SepalWidthCm 47.364461 1.327917e-16
PetalLengthCm 1179.034328 3.051976e-91
PetalWidthCm 959.324406 4.376957e-85
F-value を棒グラフにして表示します。
import matplotlib.pyplot as plt
plt.rcParams['font.size'] = 12
fig, ax = plt.subplots()
plt.bar(df_anova.index, df_anova['F-value'])
ax.tick_params(axis='x', which='major', labelsize=10)
ax.set_ylabel('F-value')
plt.grid(axis='y',linestyle='dotted')
plt.savefig('iris_041_Oneway_ANOVA_raw.png')
plt.show()
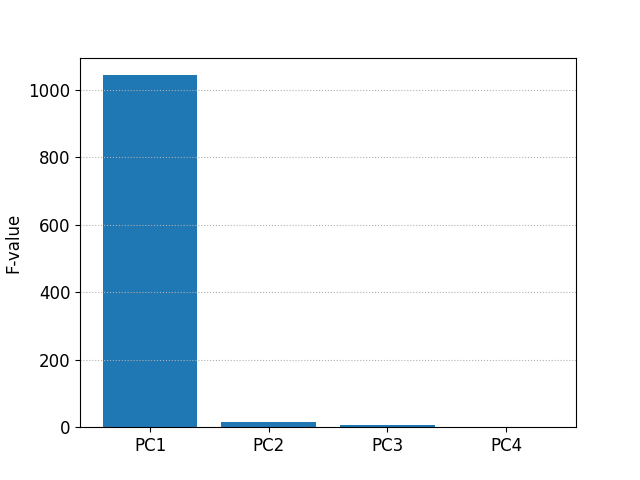
主成分の分散分析
生データと同様に、主成分 (PC1 - PC4) に対しても分散分析をします。
df_pca_anova = pd.DataFrame(columns=cols_stat)
for pc in cols_pc:
args = [df_pca[df_pca[col_y] == list_species[i]][pc] for i, species in enumerate(list_species)]
f_value, p_value = stats.f_oneway(*args)
result = pd.DataFrame(
np.array([f_value, p_value]).reshape(1, 2),
columns=cols_stat,
index=[pc]
)
# append row
df_pca_anova = pd.concat([df_pca_anova, result], axis=0)
print(df_pca_anova)
F-value p-value
PC1 1043.159859 1.412319e-87
PC2 14.429384 1.897877e-06
PC3 5.290448 6.043825e-03
PC4 1.636495 1.981874e-01
F-value を棒グラフにして表示します。
fig, ax = plt.subplots()
plt.bar(df_pca_anova.index, df_pca_anova['F-value'])
ax.set_ylabel('F-value')
plt.grid(axis='y',linestyle='dotted')
plt.savefig('iris_042_Oneway_ANOVA_PCA.png')
plt.show()
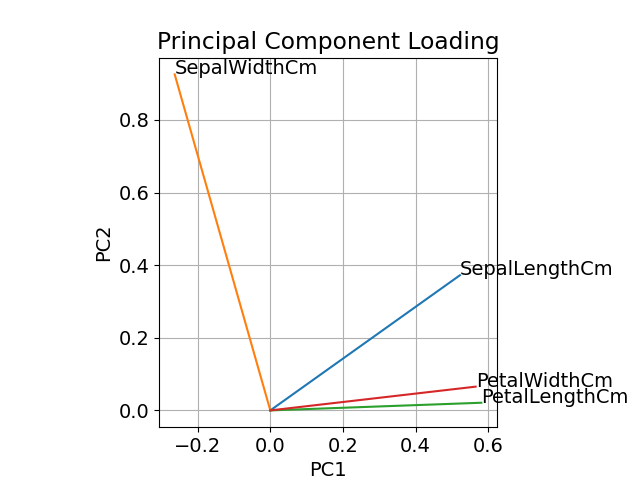
生データと主成分を分散分析した結果を比較して、あれこれ統計的な解釈をすることはできないのですが、主成分分析の結果、主成分1 (PC1) の影響がほとんどであるのに対し、生データでは PetalLengthCm(花弁の長さ)と PetalWidthCm(花弁の幅)の F-value が大きいことを考えると、Species の違いは花弁の長さと幅(= 面積?)で特徴づけられ、それは主成分分析をすると主成分1 (PC1) に現れていると考えられます。
参考までに、本ブログ記事 [1] で紹介した主成分負荷量のプロットを再掲します。
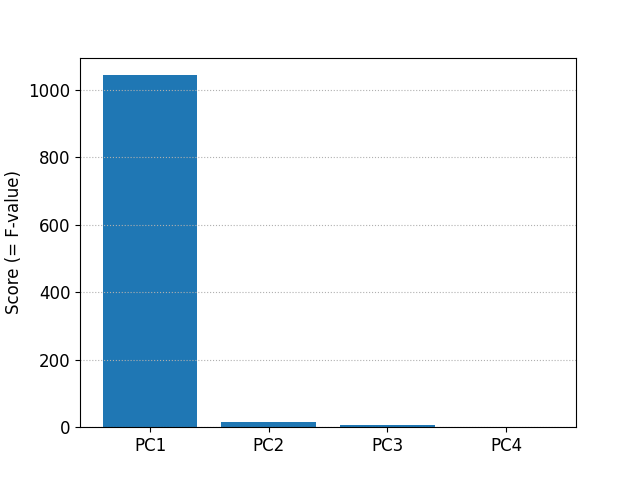
scikit-learn による分散分析
結果は同じですが、scikit-learn の SelectKBest を利用しても分散分析ができます。主成分を分散分析すると次のようになります。
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import f_classif
selector = SelectKBest(f_classif, k='all')
selector.fit(df_pca[cols_pc], df_pca[col_y])
print('features : ', selector.feature_names_in_)
print('scores : ', selector.scores_)
print('p-values : ', selector.pvalues_)
features : ['PC1' 'PC2' 'PC3' 'PC4']
scores : [1043.15985867 14.42938424 5.29044846 1.63649473]
p-values : [1.41231945e-87 1.89787708e-06 6.04382459e-03 1.98187397e-01]
score (F-value) を棒グラフにして表示します。
fig, ax = plt.subplots()
plt.bar(selector.feature_names_in_, selector.scores_)
ax.set_ylabel('Score (= F-value)')
plt.grid(axis='y',linestyle='dotted')
plt.savefig('iris_043_Feature_Selection.png')
plt.show()
参考サイト
- bitWalk's: 【備忘録】主成分分析 ~ Python / Scikit-Learn ~ [2022-12-27]
- bitWalk's: 【備忘録】主成分分析 (2) ~ Python / Scikit-Learn ~ [2023-01-03]
- bitWalk's: 【備忘録】主成分分析 (3) ~ Python / Scikit-Learn ~ [2023-01-04]


 にほんブログ村
にほんブログ村
 #オープンソース
#オープンソース